Nowości w Mnova 12
Nowy interfejs użytkownika: menu wstążkowe
Mnova 12 wprowadza całkowicie nowy interfejs użytkownika, inspirowany bardzo dobrze znanym i szeroko wykorzystywanym menu wstążkowym Windows. Jest to opcja która może być aktywowana i dezaktywowana jednym kliknięciem wg preferencji użytkownika. Zdecydowano się również zachować menu klasyczne jako menu domyślne.qGDS – poszerzenie możliwości qNMR do jego granic
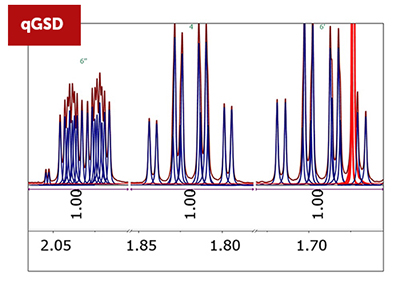
Moduł qNMR jest bardzo potężnym narzędziem analitycznym, powszechnie wykorzystywanym do opisu produktów naturalnych, analizy farmaceutycznej i kontroli jakości substancji referencyjnych. Opiera się on na dokładnym i precyzyjnym określeniu amplitud lub całek sygnału NMR, który to proces może być wykonany poprzez obliczenie sumy wszystkich punktów cyfrowych w określonym obszarze lub poprzez wykonanie technik dekonwolucyjnych.qGSD (ilościowa Globalna Dekonwolucja Widmowa) jest najnowszą innowacją wprowadzoną do Mnova, łączącą metody dekonwolucyjne celem obsłużenia sytuacji w której sygnały nachodzą na siebie.
Wersja 64-bitowa
Mnova 12 może operować nawet na największych zbiorach danych. Analiza i przetwarzanie zyskały na wydajności z możliwością użycia przetwarzania równoległego. Rozwiązane zostały również problemu z brakiem pamięci, dzięki wprowadzeniu wersji 64-bitowej Mnova.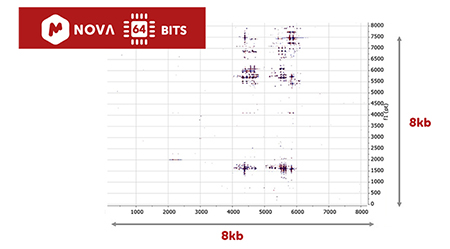

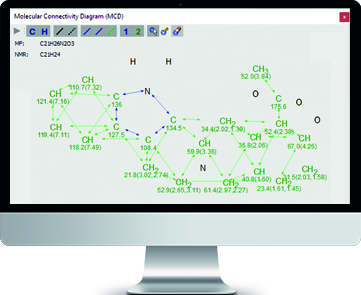
Mnova Structure Elucidator - Nowy produkt!
Narzędzie wspomagające wyjaśnianie widm NMR i wzorów cząsteczek- Przepływ od wzoru cząsteczki i danych NMR do struktury chemicznej realizowany w półautomatyczny sposób
- Łatwość i intuicyjność przepływu pracy
- Rangi struktur oparte są na odchyleniu średniokwadratowym węgla 13C
- Użycie COCON (COmputer-assisted CONstitutional assignment) jako generatora struktur

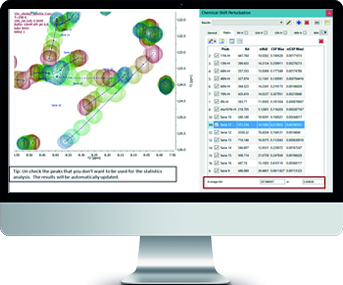
Mnova Binding - Nowy produkt!
Analiza perturbacji przesunięć chemicznych (CSP) w opracowywaniu nowych leków- W pełni automatyczny proces oraz analizy od surowych danych 2D do wartości stałych dysocjacji Kd
- Analizy związanych cząsteczek z użyciem poruszenia przesunięcia chemicznego
- Analizy manualne

Mnova IUPAC Name. Nowy produkt!
Nazwy struktur mogą być generowane na podstawie rysowanych struktur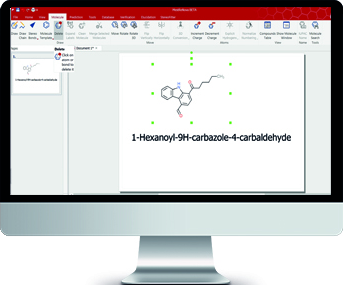

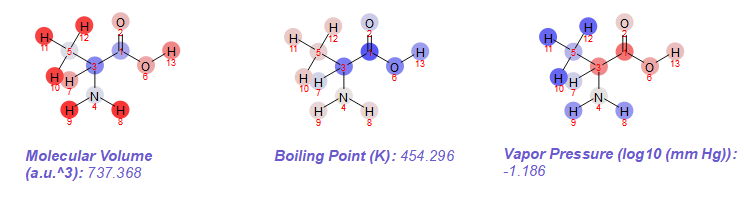
Nowości w module Mnova Physchem
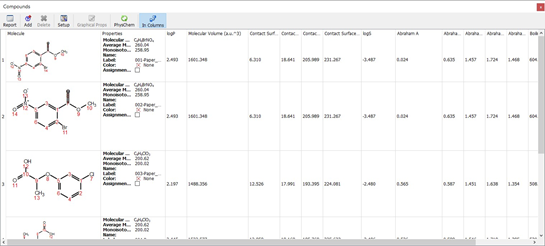
Możliwość wizualizacji atomów z użyciem schematów ich kolowania.
Automatycznie dopasowanie fazowe w analizach 1D zostało rozszerzone na analizy 2D
W wersji Mnova 12 dostępny jest nowy i algorytm automatycznej korekcji fazy dla danych NMR o wysokiej rozdzielczości. Szczegółowy opis tej funkcji można przedstawiony jest tutaj (ang).Identyfikacja wierzchołków (pików) 2D
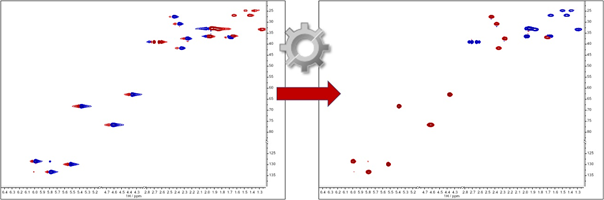
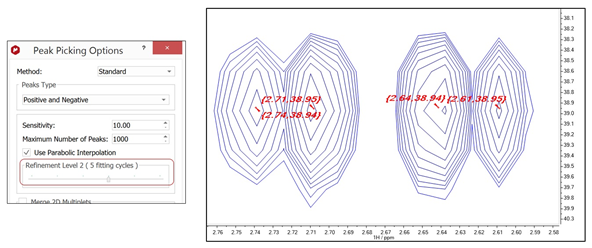
Znajdowanie pików 2D zostało znacząco ulepszone. Możliwa jest automatyczna optymalizacja parametrów, w celu uzyskania dokładniejszych wyników w przypadkach, gdy piki zachodzą na siebie.Nowe cechy w module Mnova SMA 2.0
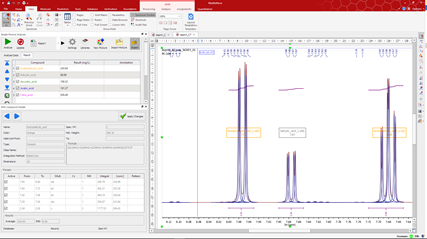
Dzięki wersji 2.0 modułu SMA, zapewnione zostało znacznie łatwiejsze określanie równań dla związków. Co ważniejsze, równanie stężenia lub czystości może być automatycznie określone przez oprogramowanie Mnova. Dodatkowo, w celu łatwiejszego określenia eksperymentu w SMA, znacznie łatwiejszym jest przegląd wyników dla pojedynczego związku lub dokonanie niezbędnych zmian. Możliwe jest na przykład wyłączenie zbioru pików (multipletu) - program automatycznie ponownie przeliczy równania stężenia i pokaże prawidłowy wynik.Inne ważne nowości oraz rozszerzenia w Mnova 12
- Dodatkowe pluginy (moduły) instalowane są obecnie bezpośrednio w programie Mnova
- Ulepszony algorytm przetwarzania MIST NUS
- Obsługa eksperymentów EXACT NMR (Craig Butts)
- Lepsza obsługa formatów plików: Jeol, Varian, Magritek, JCAMP i innych
- Możliwość importu struktur cząsteczkowych z plików JEOL
- Ulepszona dekonwolucja z możliwością użycia krzywej Gaussa
- Udoskonalenia w zadaniach